Năm 2025 là năm chúng ta chứng kiến Oxford Nanopore Technologies đạt được những bước tiến vượt trội về độ chính xác trong giải trình tự toàn bộ hệ gen vi sinh vật. Ngày càng có nhiều bằng chứng từ cộng đồng khoa học cho thấy dữ liệu nanopore, ngay cả khi sử dụng đơn lẻ, vẫn có thể đóng vai trò là công cụ then chốt cho các ứng dụng như điều tra nhanh các ổ dịch vi khuẩn, mà không cần đến NGS đọc ngắn hay các chiến lược lắp ráp lai.
Các đội ngũ của Oxford Nanopore Technologies đã nỗ lực mạnh mẽ trong việc huấn luyện các mô hình basecalling mới dành cho hệ gen vi sinh vật. Những mô hình này hiện đã cải thiện rõ rệt khả năng nhận diện các biến đổi DNA của vi khuẩn và mang lại mức tăng đáng kể về độ chính xác (bạn có thể xem một bài thuyết trình rất hay của nhóm Machine Learning của chúng tôi về cách họ huấn luyện basecaller cho các base bị biến đổi).
Kể từ đó, cộng đồng khoa học đã đưa những công cụ được cải tiến này vào thử nghiệm thực tế và nhận thấy rằng các bộ lắp ráp hệ gen được tạo ra hoàn toàn từ dữ liệu Oxford Nanopore hiện đã đạt mức độ phân giải tương đương với những gì mà chỉ vài năm trước đây còn cần phải kết hợp nhiều công nghệ giải trình tự khác nhau.
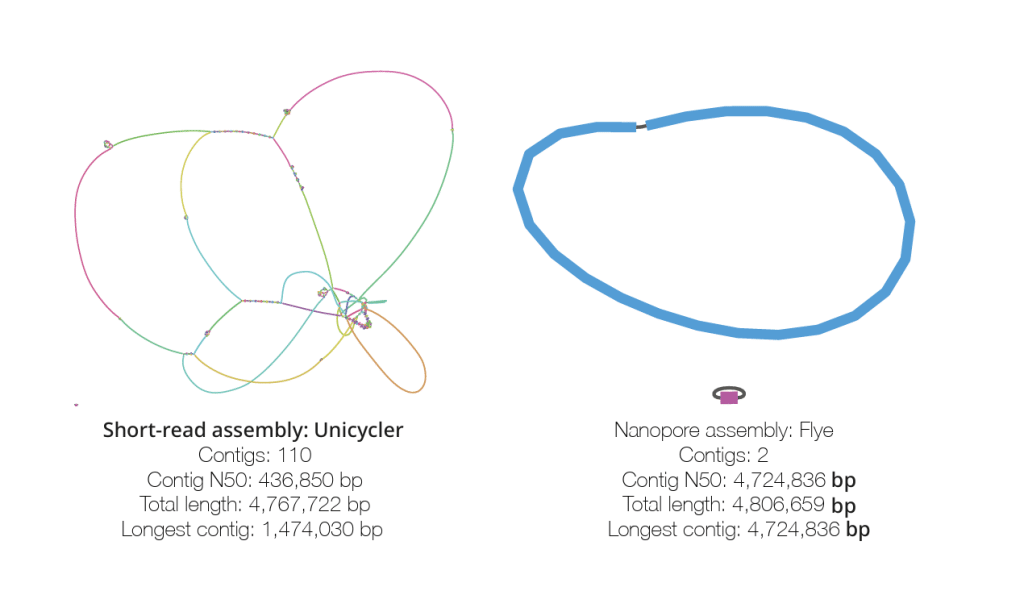
Khả năng triển khai thường quy genomics vi sinh vật trên một nền tảng duy nhất, dễ tiếp cận, mang lại:
✅ Chi phí thấp hơn
✅ Thời gian trả kết quả nhanh hơn cho các quyết định hành động
✅ Không phải đánh đổi giữa độ đầy đủ của hệ gen và độ chính xác
Dưới đây là một số trích dẫn từ các nghiên cứu gần đây thực sự nổi bật:
“Khi kết hợp độ chính xác được cải thiện của giải trình tự ONT với những lợi thế sẵn có về chi phí và phân tích, công nghệ này dường như đã sẵn sàng trở thành phương pháp chuẩn cho việc đặc trưng hóa các chủng vi khuẩn trong môi trường lâm sàng trong tương lai.”
- Nguồn: Cottingham, Hugh, et al. “Nanopore sequencing enables highly accurate genotyping and identification of resistance determinants in key nosocomial pathogens.” medRxiv (2025): 2025-07. https://www.medrxiv.org/content/10.1101/2025.07.24.25332173v1
“Sau khi tinh chỉnh các bộ lắp ráp bằng dữ liệu đọc ngắn, sai khác cặp đôi đối với cgMLST là 0 và đối với cgSNP là 0–1 SNP giữa các lần lặp. Những kết quả này cho thấy sự tiến bộ vượt bậc về độ chính xác của dữ liệu nanopore ngay cả khi không sử dụng đọc ngắn.”
- Nguồn: Abdel-Glil, Mostafa Y., et al. “High intra-laboratory reproducibility of nanopore sequencing in bacterial species underscores advances in its accuracy.” Microbial Genomics 11.3 (2025): 001372. https://www.microbiologyresearch.org/content/journal/mgen/10.1099/mgen.0.001372
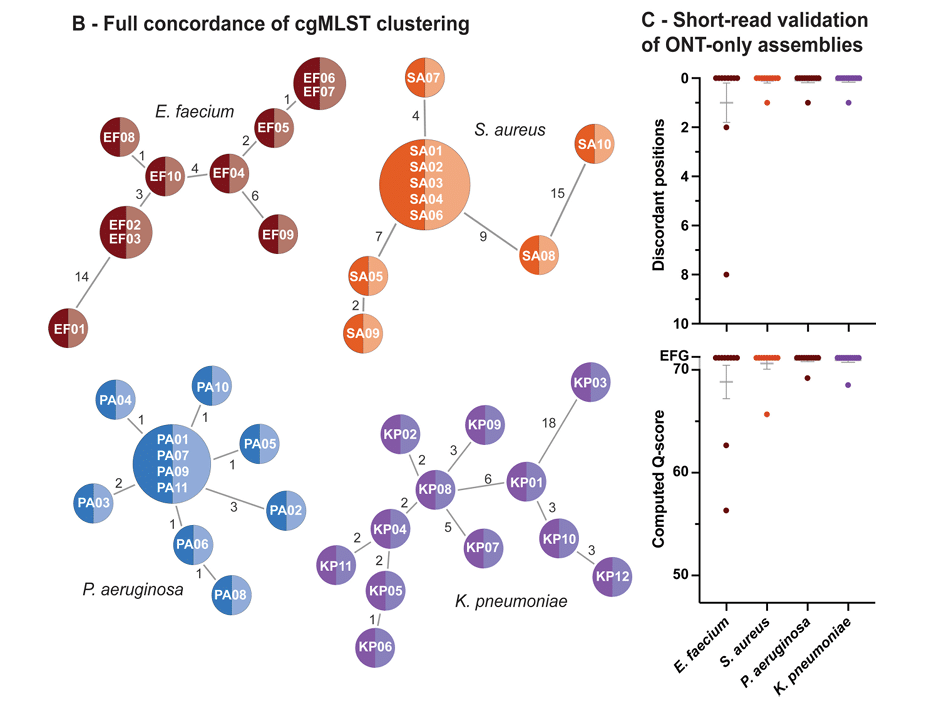
“Nghiên cứu này chứng minh rằng phân tích tính dòng (clonality) và ổ dịch chỉ dựa trên nanopore hoàn toàn khả thi, với hiệu năng tương đương giải trình tự đọc ngắn Illumina, và sẽ đóng góp quan trọng cho công tác kiểm soát nhiễm khuẩn trong bệnh viện.”
- Nguồn: Vereecke, Nick, et al. “An open-source nanopore-only sequencing workflow for analysis of clonal outbreaks delivers short-read level accuracy.” Journal of Clinical Microbiology 63.8 (2025): e00664-25. https://journals.asm.org/doi/10.1128/jcm.00664-25
“Lắp ráp hệ gen vi khuẩn chỉ sử dụng đọc dài nanopore với Autocycler kết hợp tinh chỉnh bằng Medaka cho độ chính xác tương đương, thậm chí có thể đầy đủ hơn so với lắp ráp lai, và là một giải pháp thay thế khả thi để tích hợp dữ liệu hệ gen chất lượng cao, bao gồm cả plasmid, vào giám sát Enterobacterales.”
- Nguồn: Nagy, Dorottya, et al. “Nanopore long-read only genome assembly of clinical Enterobacterales isolates is complete and accurate.” bioRxiv (2025): 2025-09. https://www.biorxiv.org/content/10.1101/2025.09.15.676237v1

Trong năm nay, chúng tôi cũng chứng kiến nhiều ca ứng dụng giám sát mầm bệnh rất ấn tượng từ các tổ chức như Eurofins và CDC PulseNet, cho thấy những tác động thực tiễn sâu rộng:
“Giải pháp của chúng tôi là triển khai phương pháp WGS chuẩn hóa của Eurofins sử dụng giải trình tự Oxford Nanopore. Kết quả rất đáng kể: nguyên nhân gốc rễ được xác định chỉ trong 5 ngày, so với đôi khi phải mất vài tuần với các phương pháp nhận diện truyền thống. Phản ứng nhanh này đã giúp tránh một đợt thu hồi sản phẩm trên toàn cầu, tiết kiệm cho công ty ước tính 15 triệu USD.” — Andrzej Benkowski, Quản lý Kỹ thuật Cấp cao, Eurofins Microbiology Laboratories
- Nguồn: Foodborne pathogen surveillance with Oxford Nanopore sequencing (YouTube)
Cuối cùng, thật đáng mừng khi thấy ngày càng nhiều nỗ lực công bố cả dữ liệu tín hiệu thô của nanopore. Những dữ liệu này cho phép re-basecalling với các mô hình trong tương lai nhằm bắt kịp các tiến bộ mới nhất, nhận diện các biến đổi DNA và motif mới để tiếp tục cải thiện độ chính xác, đồng thời nâng cao tính tái lập — chúng tôi hy vọng điều này sẽ trở thành thông lệ trong thời gian tới.
“Dữ liệu squiggle đại diện cho một kho tàng thông tin khổng lồ mà chúng ta mới chỉ bắt đầu khai thác.”
- Nguồn: Dabernig-Heinz, Johanna, et al. “A whole-genome sequencing dataset of nanopore raw signals for bacterial genotyping and methylation analysis.” Scientific Data (2025).
Hy vọng rằng trong năm 2026, chúng ta sẽ tiếp tục chứng kiến nhiều nghiên cứu xác thực hơn nữa, việc triển khai thường quy rộng rãi hơn và các ca ứng dụng có tác động lớn của giải trình tự Oxford Nanopore nhằm hỗ trợ vi sinh lâm sàng, y tế công cộng và nghiên cứu genomics trên toàn thế giới.
Tham khảo bài viết gốc: LinkedIn